本記事では、XGBoostをはじめとするGBDTモデルの基礎にある、決定木(分類) (Decision Tree Classifier) の仕組みについて、初学者にもわかりやすいように実装コードとともに説明していきます。
決定木とは?
決定木は、データを特徴に基づいて「木構造」で分類するアルゴリズムです。
- 特徴:
- 各ノード(節点)で、ある特徴量とその閾値による二分割を行う
- 葉ノード(終端ノード)では、最も多く出現するクラスラベルを予測値として採用する
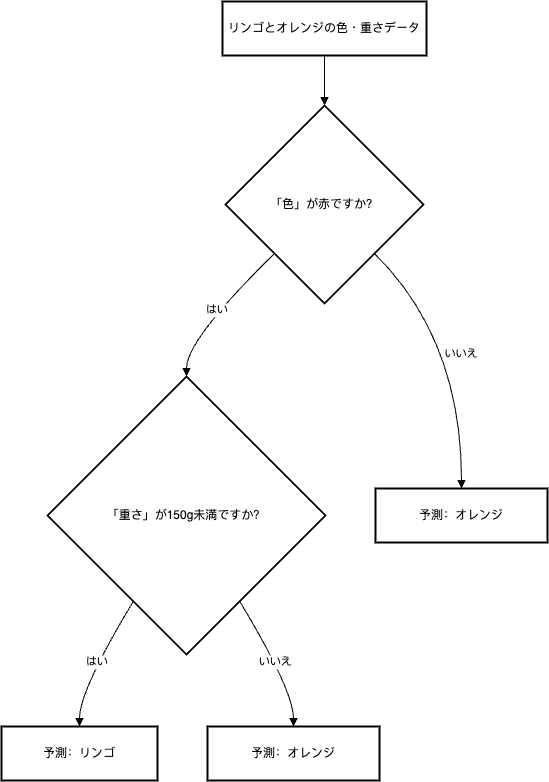
たとえば、「ある果物がリンゴかオレンジか」 を分類する場合、
- 「色」や「重さ」などの特徴により木を分岐し、最終的に「リンゴ」または「オレンジ」を予測する、といったイメージです。
数式で見る決定木の基本概念
Gini不純度
決定木の各ノードで、データの「純度」を評価するために Gini不純度 が用いられます。
Gini不純度は、あるノード内でランダムに選んだサンプルが誤分類される確率を示します。
値が小さいほど、そのノードは誤分類が少なく「純粋」であるといえます。
Gini 不純度の数式:
$$ \text{Gini} = 1 – \sum_{c=1}^{C} \left(\frac{n_c}{m}\right)^2 $$
- \( m \): ノード内のサンプル数
- \( n_c \): クラス \( c \) のサンプル数
- \( C \): クラスの総数
情報利得
情報利得は、ある分割によって Gini不純度 がどれだけ減少するかを表す指標です。
分割前の不純度から、左右の子ノードの不純度の重み付き平均を引いた値が情報利得となります。
Information Gain の数式:
$$ \text{Information Gain} = Gini_{parent} – \left(\frac{n_{left}}{n} \times Gini_{left} + \frac{n_{right}}{n} \times Gini_{right}\right) $$
- \( n \): 親ノードのサンプル数
- \( n_{left} \) と \( n_{right} \): 左右の子ノードのサンプル数
分割の候補の中で情報利得が最大になるものを選び、最適な分割とします。
実装の流れ
決定木の実装は、概ね以下の流れで進みます。
- ノードの定義
各ノードを表現するクラスを作成します。ここでは、分割に用いる特徴量のインデックスや閾値、左右の子ノード、または葉ノードの予測値を保持します。 - 再帰的な木の成長
トップノードから始め、条件(最大深度、サンプル数、同一クラスなど)を満たすまで、再帰的にノードを分割していきます。
各ノードで最適な特徴量と閾値の組み合わせを探索し、左右に分けます。 - 予測処理
新しいサンプルに対して、ルートから始めて各ノードの条件を順に確認し、最終的にたどり着いた葉ノードの予測値を返します。
部分的な実装コード
以下は、上記の各要素に対応するコードの一部です。
Node クラス
from collections import Counter
# Node class
class Node:
def __init__(self, feature_index=None, threshold=None, left=None, right=None, *, value=None):
# Index of feature used for split
self.feature_index = feature_index
# Threshold value for split
self.threshold = threshold
# Left child node
self.left = left
# Right child node
self.right = right
# Predicted class for leaf node (if not None, node is a leaf)
self.value = value
def is_leaf_node(self):
return self.value is not NoneDecisionTreeClassifier クラス(主要部分)
import numpy as np
# Custom Decision Tree Classifier implementation
class DecisionTreeClassifier:
def __init__(self, min_samples_split=2, max_depth=100, n_features=None):
# Minimum number of samples required to split a node
self.min_samples_split = min_samples_split
# Maximum depth of the tree
self.max_depth = max_depth
# Number of features to consider when looking for the best split (if None, use all)
self.n_features = n_features
self.root = None
# Fit the model on the training data
def fit(self, X, y):
self.n_classes_ = len(set(y))
self.n_features_ = X.shape[1]
if self.n_features is None:
self.n_features = self.n_features_
self.root = self._grow_tree(X, y)
# Recursively build the tree
def _grow_tree(self, X, y, depth=0):
n_samples, n_features = X.shape
n_labels = len(np.unique(y))
# Stop criteria: max depth reached, all samples have the same label, or not enough samples to split
if (depth >= self.max_depth or n_labels == 1 or n_samples < self.min_samples_split):
leaf_value = self._most_common_label(y)
return Node(value=leaf_value)
# Randomly select features (if n_features is less than total number of features)
feature_idxs = np.random.choice(n_features, self.n_features, replace=False)
# Find the best split among the selected features
best_feat, best_thresh = self._best_split(X, y, feature_idxs)
if best_feat is None:
leaf_value = self._most_common_label(y)
return Node(value=leaf_value)
# Split the data based on the best feature and threshold
left_idxs = X[:, best_feat] <= best_thresh
right_idxs = X[:, best_feat] > best_thresh
# Recursively build left and right subtrees
left = self._grow_tree(X[left_idxs], y[left_idxs], depth + 1)
right = self._grow_tree(X[right_idxs], y[right_idxs], depth + 1)
return Node(feature_index=best_feat, threshold=best_thresh, left=left, right=right)
# Determine the best split by calculating information gain using Gini impurity
def _best_split(self, X, y, feature_idxs):
best_gain = -1
split_idx, split_thresh = None, None
for feat_idx in feature_idxs:
X_column = X[:, feat_idx]
thresholds = np.unique(X_column)
for threshold in thresholds:
gain = self._information_gain(y, X_column, threshold)
if gain > best_gain:
best_gain = gain
split_idx = feat_idx
split_thresh = threshold
return split_idx, split_thresh
# Calculate information gain using the decrease in Gini impurity
def _information_gain(self, y, X_column, split_thresh):
parent_gini = self._gini(y)
left_idxs = X_column <= split_thresh
right_idxs = X_column > split_thresh
# If either split is empty, no information gain is achieved
if len(y[left_idxs]) == 0 or len(y[right_idxs]) == 0:
return 0
n = len(y)
n_left, n_right = len(y[left_idxs]), len(y[right_idxs])
gini_left, gini_right = self._gini(y[left_idxs]), self._gini(y[right_idxs])
weighted_gini = (n_left / n) * gini_left + (n_right / n) * gini_right
gain = parent_gini - weighted_gini
return gain
# Compute the Gini impurity for a set of labels
def _gini(self, y):
m = len(y)
return 1.0 - sum((np.sum(y == c) / m) ** 2 for c in np.unique(y))
# Return the most common label in the set
def _most_common_label(self, y):
from collections import Counter
counter = Counter(y)
most_common = counter.most_common(1)[0][0]
return most_common
# Predict the class for each sample in X
def predict(self, X):
return np.array([self._traverse_tree(x, self.root) for x in X])
# Recursively traverse the tree to make a prediction
def _traverse_tree(self, x, node):
if node.is_leaf_node():
return node.value
if x[node.feature_index] <= node.threshold:
return self._traverse_tree(x, node.left)
return self._traverse_tree(x, node.right)完成版のコード
以下は、上記の各部分を統合した、完成版のカスタム DecisionTreeClassifier のコードです。
また、軽量なデータセット(ここでは Iris データセット)を用いて、実際に学習・予測を行う例も含めています。
import numpy as np
import pandas as pd
from collections import Counter
from sklearn.datasets import load_iris
from sklearn.model_selection import StratifiedKFold
from sklearn.metrics import accuracy_score
from tqdm import tqdm
# ---------------------------
# Node class representing a tree node
# ---------------------------
class Node:
def __init__(self, feature_index=None, threshold=None, left=None, right=None, *, value=None):
# Index of feature used for split
self.feature_index = feature_index
# Threshold value for split
self.threshold = threshold
# Left child node
self.left = left
# Right child node
self.right = right
# Predicted class for leaf node (if not None, node is a leaf)
self.value = value
def is_leaf_node(self):
return self.value is not None
# ---------------------------
# Custom Decision Tree Classifier implementation
# ---------------------------
class DecisionTreeClassifier:
def __init__(self, min_samples_split=2, max_depth=100, n_features=None):
# Minimum number of samples required to split a node
self.min_samples_split = min_samples_split
# Maximum depth of the tree
self.max_depth = max_depth
# Number of features to consider when looking for the best split (if None, use all)
self.n_features = n_features
self.root = None
# Fit the model on the training data
def fit(self, X, y):
self.n_classes_ = len(set(y))
self.n_features_ = X.shape[1]
if self.n_features is None:
self.n_features = self.n_features_
self.root = self._grow_tree(X, y)
# Recursively build the tree
def _grow_tree(self, X, y, depth=0):
n_samples, n_features = X.shape
n_labels = len(np.unique(y))
# Stop criteria: max depth reached, all samples have the same label, or not enough samples to split
if (depth >= self.max_depth or n_labels == 1 or n_samples < self.min_samples_split):
leaf_value = self._most_common_label(y)
return Node(value=leaf_value)
# Randomly select features (if n_features is less than total number of features)
feature_idxs = np.random.choice(n_features, self.n_features, replace=False)
# Find the best split among the selected features
best_feat, best_thresh = self._best_split(X, y, feature_idxs)
if best_feat is None:
leaf_value = self._most_common_label(y)
return Node(value=leaf_value)
# Split the data based on the best feature and threshold
left_idxs = X[:, best_feat] <= best_thresh
right_idxs = X[:, best_feat] > best_thresh
# Recursively build left and right subtrees
left = self._grow_tree(X[left_idxs], y[left_idxs], depth + 1)
right = self._grow_tree(X[right_idxs], y[right_idxs], depth + 1)
return Node(feature_index=best_feat, threshold=best_thresh, left=left, right=right)
# Determine the best split by calculating information gain using Gini impurity
def _best_split(self, X, y, feature_idxs):
best_gain = -1
split_idx, split_thresh = None, None
for feat_idx in feature_idxs:
X_column = X[:, feat_idx]
thresholds = np.unique(X_column)
for threshold in thresholds:
gain = self._information_gain(y, X_column, threshold)
if gain > best_gain:
best_gain = gain
split_idx = feat_idx
split_thresh = threshold
return split_idx, split_thresh
# Calculate information gain using the decrease in Gini impurity
def _information_gain(self, y, X_column, split_thresh):
parent_gini = self._gini(y)
left_idxs = X_column <= split_thresh
right_idxs = X_column > split_thresh
# If either split is empty, no information gain is achieved
if len(y[left_idxs]) == 0 or len(y[right_idxs]) == 0:
return 0
n = len(y)
n_left, n_right = len(y[left_idxs]), len(y[right_idxs])
gini_left, gini_right = self._gini(y[left_idxs]), self._gini(y[right_idxs])
weighted_gini = (n_left / n) * gini_left + (n_right / n) * gini_right
gain = parent_gini - weighted_gini
return gain
# Compute the Gini impurity for a set of labels
def _gini(self, y):
m = len(y)
return 1.0 - sum((np.sum(y == c) / m) ** 2 for c in np.unique(y))
# Return the most common label in the set
def _most_common_label(self, y):
counter = Counter(y)
most_common = counter.most_common(1)[0][0]
return most_common
# Predict the class for each sample in X
def predict(self, X):
return np.array([self._traverse_tree(x, self.root) for x in X])
# Recursively traverse the tree to make a prediction
def _traverse_tree(self, x, node):
if node.is_leaf_node():
return node.value
if x[node.feature_index] <= node.threshold:
return self._traverse_tree(x, node.left)
return self._traverse_tree(x, node.right)
# ---------------------------
# Demonstration with the Iris dataset
# ---------------------------
if __name__ == '__main__':
# Load the Iris dataset
iris = load_iris()
X = iris.data
y = iris.target
# Use Stratified K-Fold cross-validation for demonstration
folds = 5
skf = StratifiedKFold(n_splits=folds, shuffle=True, random_state=42)
oof_preds = np.zeros(len(X))
test_preds = np.zeros(len(X)) # For demonstration, we use the same data as test
# Cross-validation loop with a progress bar
for fold, (train_idx, val_idx) in enumerate(tqdm(list(skf.split(X, y)), total=folds, desc="Folds")):
print(f"\nFold {fold+1}/{folds}")
X_train, y_train = X[train_idx], y[train_idx]
X_val, y_val = X[val_idx], y[val_idx]
# Create and train the custom Decision Tree Classifier
model = DecisionTreeClassifier(max_depth=5, min_samples_split=2)
model.fit(X_train, y_train)
# Predict on the validation set
val_preds = model.predict(X_val)
oof_preds[val_idx] = val_preds
acc = accuracy_score(y_val, val_preds)
print(f"Fold {fold+1} Accuracy: {acc:.4f}")
# For demonstration, assign validation predictions as test predictions
test_preds[val_idx] = model.predict(X_val)
overall_acc = accuracy_score(y, oof_preds)
print(f"\nOverall Accuracy: {overall_acc:.4f}")
print("Test Predictions:", test_preds)まとめ
本記事では、決定木(分類)の基本概念、数学的背景、そして実装の流れを詳しく解説しました。
もし本記事の内容に誤りや改善点があれば、コメントいただければ幸いです。
付録: 回帰の場合
回帰木における分割基準(分散の減少)の数式:
$$ \text{Reduction} = \text{Var}_{parent} – \left(\frac{n_{left}}{n} \times \text{Var}_{left} + \frac{n_{right}}{n} \times \text{Var}_{right}\right) $$
- \( n \): 親ノードのサンプル数
- \( n_{left} \) と \( n_{right} \): 左右の子ノードのサンプル数
- \( \text{Var}_{parent} \), \( \text{Var}_{left} \), \( \text{Var}_{right} \): それぞれ親ノード、左ノード、右ノードの分散
分類木との違い:
分類木では、各ノードの純度を評価するためにGini不純度やエントロピーを用い、情報利得(不純度の減少)を基準として分割を行います。一方、回帰木では、目的変数の分散(または平均二乗誤差)の減少量を評価基準とし、分割前後の分散の差(分散の減少)が最大になるような分割を選びます。



コメント